Neue Bakterienart im Darm entdeckt
Bio-News vom 29.09.2022
Ob Pflanze, Tier oder Mensch, lebende Organismen sind von einer Vielzahl an Bakterien besiedelt. Forschungsergebnisse der letzten Jahre zeigen, dass Bakterien mit ihrem Wirt nicht nur koexistieren, sondern wechselseitige Interaktionen in Form einer Symbiose eingehen, die sowohl dem Bakterium als auch dem Wirt von Nutzen sein kann.
Diese funktionelle Zusammenarbeit zwischen Bakterien und dem Wirt wird von Wissenschaftlerinnen und Wissenschaftlern als Metaorganismus (auch Holobiont) bezeichnet. Dessen Evolutionsgeschichte und Funktion untersuchen Forschende von der Christian-Albrechts-Universität zu Kiel (CAU) und dem Max-Planck-Institut für Evolutionsbiologie in Plön (MPI-EB) unter anderem im DFG-Sonderforschungsbereich (SFB) 1182 „Entstehen und Funktionieren von Metaorganismen“.
Publikation:
Hanna Fokt, Rahul Unni, Urska Repnik, Ruth A Schmitz, Marc Bramkamp, John F Baines, Daniel Unterweger
Bacteroides muris sp. nov. isolated from the cecum of wild-derived house mice
Archives of Microbiology 204(9):546 (2022)
DOI: 10.1007/s00203-022-03148-6
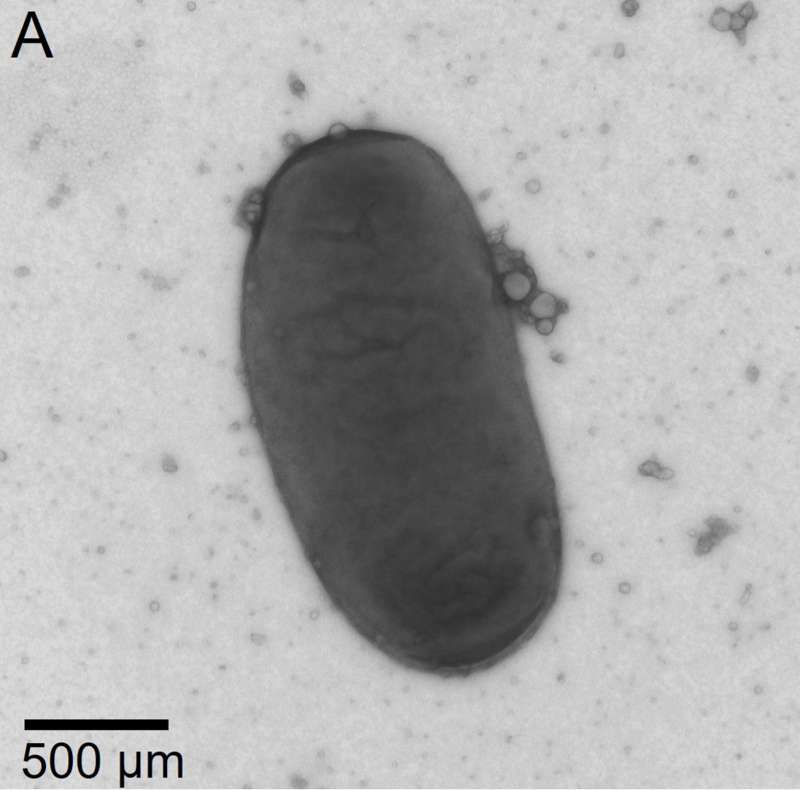
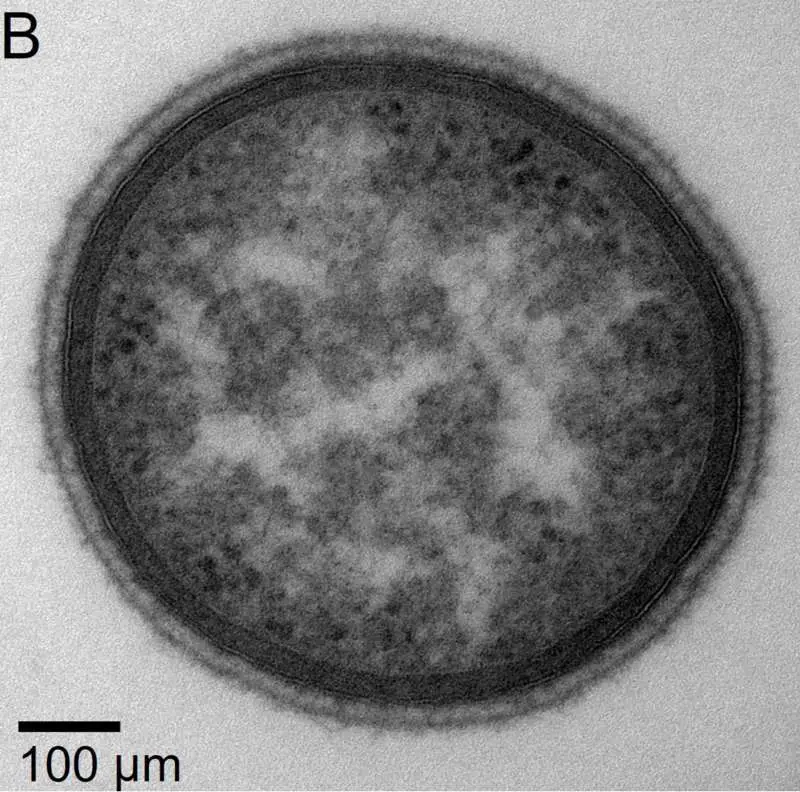
Bacteroides-Bakterien kommen in großer Anzahl in der Mikrobiota von Säugetieren und auch Menschen vor. Mit den beiden kürzlich neu entdeckten Stämmen, die aus dem Blinddarm von wildlebenden Hausmäusen in der Tieranlage des MPI-EB isoliert wurden, erhofft sich die Forschungsgruppe Evolutionsmedizin nun neue Erkenntnisse zu erzielen. „Ein Ziel ist es mit dieser Entdeckung langfristig besser zu verstehen, warum Bakterien auf und in uns leben. Manche dieser Bakterien sind noch immer unbekannt. Jetzt haben wir sehr grundlegende Erkenntnisse über diese Bakterienart und wissen beispielsweise von Aufnahmen mit dem Mikroskop wie diese Bakterien aussehen und welche Größe sie haben“, sagt Dr. Daniel Unterweger, Forschungsgruppenleiter am MPI für Evolutionsbiologie und an der CAU, sowie Mitglied im SFB 1182.
Die Entdeckung kam unerwartet. Um die Identität neu isolierter Bakterien zu bestimmen, wird mit modernen Sequenziermethoden das bakterielle Genom entschlüsselt und mit bekannten Bakterienarten abgeglichen. „Es war eine große Überraschung, dass die von uns isolierten Bakterien mit keiner bekannten Bakterienart in der Datenbank übereinstimmten und entsprechend noch keinen Namen hatten“, beschreibt Erstautorin Dr. Hanna Fokt, Postdoktorandin am MPI-EB und Wissenschaftlerin im SFB 1182, den besonderen Moment, als klar wurde, eine neue Bakterienart entdeckt zu haben.
Die Ergebnisse anschließender genomischer und phänotypischer Analysen deuten darauf hin, dass die beiden hier beschriebenen Stämme eine neue Art der Gattung Bacteroides darstellen. Diese neuen Bakterienstämme könnten nicht nur bei Mäusen, sondern auch bei Menschen vorkommen und versprechen daher ein besseres Verständnis der Evolution der Gattung Bacteroides als wichtiges Mitglied einer gesunden Säugetiermikrobiota. Die Zusammensetzung dieser Mikrobiota und seine Interaktionen mit Wirtszellen stehen im Mittelpunkt des Forschungsinteresses im SFB 1182.
Die erste Bakterienart der Bacteroides wurde Ende des 19. Jahrhunderts beschrieben und trägt den Namen Bacteroides fragilis. Seitdem wurden insgesamt 35 verschiedene Bacteroides-Arten isoliert, charakterisiert und validiert. Verbesserte technische Möglichkeiten der Genomsequenzierung ermöglichen es heute, die Verwandtschaft von Bakterien sehr genau zu bestimmen. Aufbewahrt werden Kulturen der Bakterienarten unter anderem in der Deutschen Sammlung für Mikroorganismen und Zellkulturen in Braunschweig und sind dadurch Forschenden weltweit zugänglich.
Diese Newsmeldung wurde mit Material des Max-Planck-Instituts für Evolutionsbiologie via Informationsdienst Wissenschaft erstellt.